小白刷题之 187 ~ 重复的 DNA 序列
基本信息
- 题号:187
- 题厂:脸书
- 难度系数:中
解题思路
- 为了找出字符串中出现过 2+ 次数 且 长度 == 10 的 substring,引用 set 记录出现过的 substring。
- 这种比较 substring 之类的题,一般思路都是 sliding window。
只要想到了 sliding window + set,本题引刃而解。
class Solution:
def findRepeatedDnaSequences(self, s: str) -> List[str]:
# 当输入字符串长度小于、等于 10 时,是不可能找到出现 2+ 次 且 长度 == 10 的 substring。可以提前返回空数组
if len(s) <= 10:
return []
# 当输入字符串长度大于 10 时,才有可能找到重复的 DNA 序列。用 seq 记录出现过的所有 长度 == 10 的 substring。
# 当在遍历过程中,遇上重复出现的 substring,将它添加进 res。 遍历完成后,返回 res 即要找的结果。
seq = {s[:10]}
res = set()
# 做 for 循环时,为了保证数组不越界(始终有 长度 == 10 的substring 进行比较),起点为 1,终点为 输入字符串长度剪 9
for r in range(1, len(s) - 9):
cur = s[r: r + 10]
if cur in seq:
res.add(cur)
else:
seq.add(cur)
# 返回前,需要把 set 换回 list,带到避免返回结果重复的目的。
return list(res)
Constraints
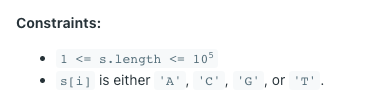
获得最小字符串长度可以是 1,所以需要对字符串长度小于 10 的案例进行特殊处理。
考试时,讨论解题思路准备写代码前,一定要向考官确认输入字符串最大长度和最小长度。
Big O
- Time:对 s 进行了一次 for 循环遍历,因此复杂度为 O(n)。其中 n 为 输入字符串 s 的长度。
- space:使用了 set,于是复杂度为 O(n)。最坏情况下,一直遇上不同 substring,不断往 seq 里吗添加。。。。
测试
除了当 s 长度小于 10 时,还可以重点测试字符串长度 == 11 时的案例。
总结
此题为脸书偏简单的题。因为涉及的都是基本数据结构,难度系数属于中档偏简单。只要熟悉 sliding window 原理,考试遇上此类型应在 20 分钟之内解答。
注意检查如何保证返回数组无重复,和细致处理数组边界问题。